DMF - Erforschung und Identifizierung aquatischer Pilze mit einer Kombination aus Mikroskopie und molekularen Werkzeugen
Pilze sind als wesentlicher Bestandteil sowohl terrestrischer als auch aquatischer Ökosysteme ein wichtige Gruppe von Mikroorganismen. Sie spielen eine entscheidende Rolle beim Abbau organischer Stoffe und beim Nährstoffkreislauf und ihre Vielfalt wird inzwischen auf 2,2 bis 3,8 Millionen Arten geschätzt. Viele davon sind jedoch noch unbekannt, denn es gibt nur etwa 149 000 formal beschriebene Pilzarten. Diese Diskrepanz zwischen bekannten und unbekannten Pilzen ist in aquatischen Lebensräumen noch ausgeprägter, da nur eine Handvoll aquatischer Pilze entweder isoliert oder charakterisiert ist. Noch komplizierter wird es, wenn es um frühe Pilze geht, d.h. Microsporidia, Cryptomycota, Chytridiomycota, da es fast keine Vertreter in Referenzkulturen oder Sequenzdatenbanken gibt und die meisten beschriebenen Arten auf Zeichnungen beruhen. Um diese Lücke zwischen bekannten und unbekannten Pilzen zu schließen, wurden kulturunabhängige Sequenzierungstechnologien der nächsten Generation eingesetzt. Diese Bemühungen reichten jedoch nicht aus, um den Fungal Tree of Life (FToL) mit neu beschriebenen Pilzarten zu füllen.
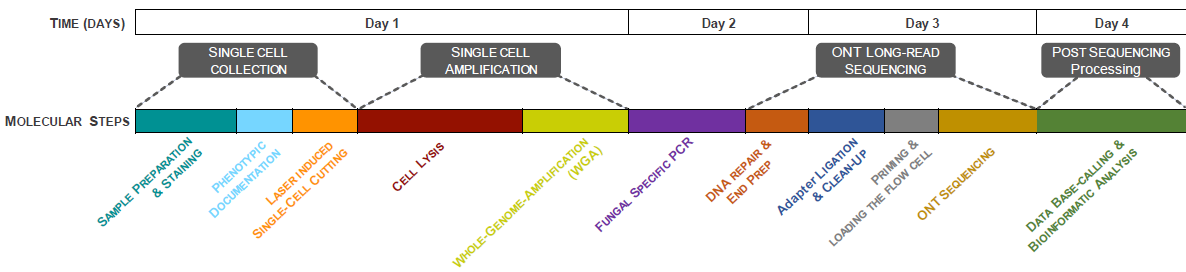
Daher hat sich die Microbial System Research Group zum Ziel gesetzt, eine Toolbox zu entwickeln, um dieses zentrale Problem der Dark Matter Fungi (DMF) in aquatischen Lebensräumen zu lösen. Zu diesem Zweck arbeiten wir mit einzelnen Pilzzellen unter Verwendung des Laser Dissection Microscope (LMD7) in Kombination mit einem Long-Read-Sequenzer der dritten Generation (Oxford Nanopore) (Abbildung). Durch die Kombination der Laser-Dissektion einzelner Pilzzellen mit der vollständigen Amplifikation des ribosomalen Operons von Pilzen schaffen wir eine methodische Plattform (Eco-ACDCS: Ecological Annotated Cell Dissection for Cultivation and nucleic acid Sequencing), die der Schlüssel zur Aufklärung der dunklen Materie von Pilzen in aquatischen Lebensräumen sein wird.
Mit der Realisierung der Eco-ACDCS-Plattform werden wir die unbekannten Pilzlinien in unserer Umwelt systematisch katalogisieren, mit dem Ziel:
- die Identität von aquatischen Pilzen zu erfassen, insbesondere bei der Arbeit mit DNA-Sequenzdaten
- die Phylogenie neuartiger Pilzstämme durch die Verwendung mehrerer Marker oder durch den Übergang zu einzelnen Zellen zu klären
- der Durchführung von Hochdurchsatzanalysen von Süßwasserpilzen aus unterschiedlichen Umweltproben
- eine Brücke zwischen der Vergangenheit und der Zukunft zu schlagen, indem Referenzen für aquatische Pilze bereitgestellt werden
- Bereitstellung einer referenzierten DNA-Bank für Einzelzellen und anschließende Genomanalyse
Mit der Eco-ACDCS-Plattform werden wir eine wichtige Rolle dabei spielen, die aquatische Mykologie vom Unbekannten zum Bekannten voranzutreiben, insbesondere für früh divergierende Pilzlinien. Unsere Referenzsequenzen und die DNA-Bank für einzelne Pilzzellen werden die zukünftige Forschung weiter erleichtern.

Daher haben wir als Teil der Microbial System Research Group erfolgreich einen Arbeitsablauf etabliert, der die Laser-Mikrodissektion einzelner Pilzzellen mit der Amplifikation ganzer Genome und der Long-Read-Sequenzierung kombiniert. Wir haben verschiedene aquatische Lebensräume auf die Jagd nach bestimmten Pilzgruppen getestet, z. B. aquatische Hyphomyceten (bekannt für den Abbau von Laubstreu) und Chytriden (parasitische Pilze) - mit Erfolg. Die Erfolgsquote liegt zurzeit bei 59 % für die Amplifikation der kurzen ITS-Region und bei 24 % für die langen ribosomalen Operonregionen der rDNA. Dieser Arbeitsablauf ermöglichte es uns, die taxonomische und phylogenetische Einordnung einer unbekannten Pilzart innerhalb von einer Woche zu bestimmen (Abbildung 2).
Darüber hinaus haben wir die Anwendung unseres Arbeitsablaufs auf die aquatischen Lebensräume in den arktischen Regionen Nordkanadas und die extremen Gewässer Islands ausgeweitet. Im Sommer 2022 haben wir in den arktischen Gebieten Nordkanadas Proben aus Permafrost-Tauweihern unterschiedlichen Alters (Abbildung 3a) und aus den Gletscherseen und heißen Quellen Islands (Abbildung 3b) genommen. Die Proben werden aufbereitet und werden dazu beitragen, die unbekannten Pilzlinien aus solchen Umgebungen zu erschließen, die unter dem Gesichtspunkt der globalen Erwärmung und der extremophilen Mykologie von Bedeutung sind.

|
| |
| Projektleiter | Dr. Christian Wurzbacher |
| Sachbearbeiter | |
| Förderung | Deutsche Forschungsgemeinschaft - DFG |


